踵事增华 | 为什么原位敲入细胞系构建前,一定要做蛋白表达验证?
2025-09-10
在生命科学研究中,CRISPR/Cas9 介导的原位插入(Endogenous Knock-in)技术堪称 “利器”——它能在目标基因的内源性启动子调控下精准插入标签,让蛋白表达、定位和动态变化 “可视化”,为解析生命机制提供关键支持。
但这项技术也藏着一个容易被忽略的“隐形坑”:如果跳过目的基因内源表达验证,哪怕基因组插入成功,后续实验也可能功亏一篑。今天就通过一个真实案例,聊聊表达验证的重要性。
一、什么是原位插入?为什么它这么重要?
原位插入技术通过CRISPR/Cas9 系统,将标签(如 EGFP、HiBiT)精准插入目标基因的特定位置(比如 C 端),让蛋白在自身启动子调控下表达。这种方式的核心优势在于:
-
保留天然状态:蛋白表达水平、活性和细胞内定位与天然状态一致,避免外源启动子带来的“过度表达干扰”;
-
可视化追踪:通过标签可直接观察蛋白的亚细胞定位、动态变化,甚至互作蛋白;
-
稳定性强:标签随基因稳定遗传,适合长期实验。
正因如此,它成为研究蛋白功能、相互作用的核心工具。但它有个“软肋”:高度依赖目的基因的内源表达水平。如果基因本身在细胞中表达量极低,就算插入成功,标签也可能 “沉默”,后续根本检测不到信号。
二、真实案例:插入成功,却测不到信号?
我们以“Htert-RPE1 细胞中给 R 基因 C 端插入 EGFP-HiBiT 标签” 为例,看看问题出在哪。
2.1 第一步:基因组验证显示“插入成功”
实验设计很标准:在R 基因 C 端设计 sgRNA,用 CRISPR/Cas9 系统插入 EGFP-HiBiT 标签。通过 Sanger 测序验证,结果显示标签已精准插入 R 基因(如图 1)—— 从基因组层面看,插入完全成功。(如图1)。
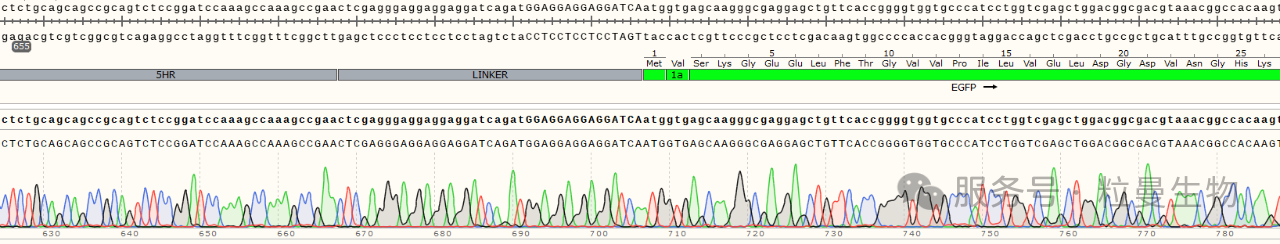
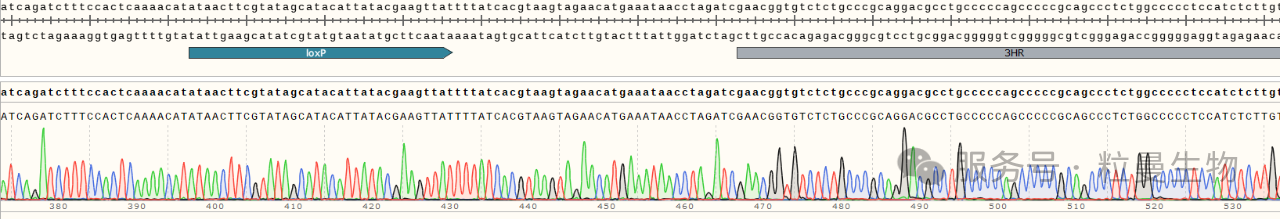
1 Htert-RPE1-R(C-GFP)sanger测序结果
2.2 第二步:表达验证却“全军覆没”
本以为接下来的蛋白检测会很顺利,但结果却出人意料:
1)R 蛋白抗体检测:用 R 蛋白抗体做 Western blot(WB),既没检测到带标签的 “R-GFP 蛋白”,连天然 R 蛋白的条带也模糊不清(如图 2)。
2)GFP 抗体检测:换 GFP 抗体检测标签,依然没有明显条带(如图 3)。
两种方法都失败,核心原因指向一个:R 基因在 Htert-RPE1 细胞中内源表达量太低—— 就像 “在安静的房间里说话,音量太小就传不出去”,低表达的蛋白连带着标签也难以被检测到。
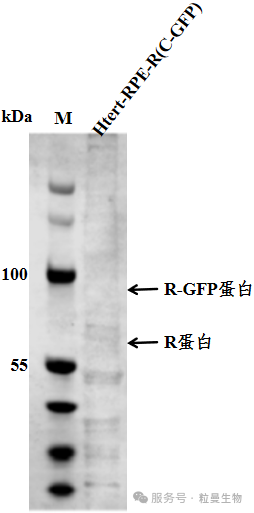
图2 R蛋白抗体WB结果
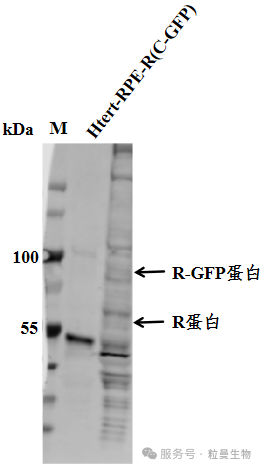
图3 GFP抗体WB结果
2.3 第三步:高灵敏度标签“救场”
好在我们同时插入了HiBiT 标签(一种仅11个氨基酸的小肽,检测灵敏度极高)。用 HiBiT 系统检测发现:
-
相比空白对照,插入标签的细胞有明显信号(如图4),证明插入的标签确实在表达;
-
但信号强度仅为阳性对照的1/20,进一步证实 R 基因在该细胞中表达量极低。
这个结果也说明:如果只依赖传统标签(如EGFP)和 WB 检测,很可能误以为 “插入失败”,甚至放弃正确的细胞系。
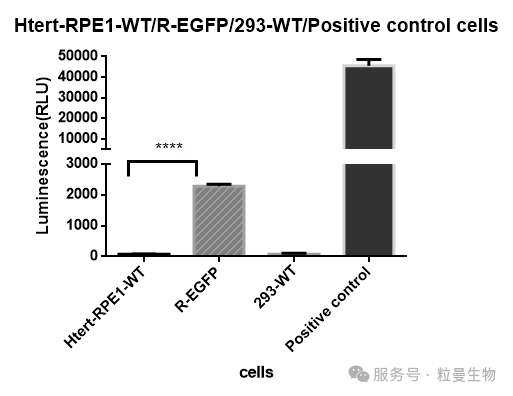
图4 Htert-RPE1-R(C-GFP)的HiBiT表达情况
关键结论:表达验证,是避坑的核心
这个案例告诉我们:
基因组插入成功≠蛋白能被检测到。如果跳过 “内源表达验证”,可能出现两种后果:
-
白做实验:花时间构建细胞系,却因检测不到信号而浪费资源;
-
误判结果:把“低表达导致的无信号” 当成 “插入失败”,错过正确方向。
三、实操建议:3 步避坑指南
基于大量项目经验,我们总结了原位插入前的“必做评估步骤”,帮你少走弯路:
1. 先查内源表达水平
用WB检测目标基因在目标细胞中的天然表达量——如果本身表达极低,提前预警 “后续检测风险”。
2. 选对细胞系
如果目标细胞表达太低,可换成该基因高表达的细胞系(如HEK293T、Hela 等常见模型),从源头提高成功率。
3. 用高灵敏度标签
对低表达蛋白,放弃传统GFP 标签,改用 HiBiT 或 Nanoluc 荧光素酶标签:
-
它们的检测灵敏度比WB 高 100-1000 倍,能捕捉到微弱信号;
-
小肽标签(如HiBiT)对蛋白功能影响更小,更适合低表达基因。
四、写在最后
原位插入技术是研究的“加速器”,但只有做好前期准备,才能让它真正发挥作用。内源表达验证不是“额外步骤”,而是避免无效投入的关键。如果你在原位插入细胞系构建中遇到过类似问题,或有相关需求,欢迎留言交流——我们踩过的坑,或许能帮你少走一些路。
(注:文中案例及数据均来自粒曼生物实验结果,如需进一步技术细节,可联系我们获取完整方案。)
2025 /
09-10
所属分类:
行业新闻
相关资讯—
2025-12-08
2025-12-08
2025-12-08

