技术专题 | 邻近标记:蛋白互作研究的“时空穿梭机”——从BioID到TurboID的技术演进与应用【收藏】
2025-08-25

在细胞生物学研究中,揭示蛋白质相互作用、RNA-蛋白质相互作用及细胞间相互作用是理解生命活动的关键。近年来,邻近标记技术(Proximity Labeling) 的兴起为这些问题提供了高效、精准的解决方案。本文将从技术发展史出发,梳理邻近标记技术的演进历程,并总结各技术的优缺点,为科研工作者提供参考。
一、历史沿革:从“慢标记” 到 “精准时空控制” 的技术突破
1. BioID(2012):开启邻近标记时代的 “慢而稳” 工具
-
开发者:Brian Burke实验室
-
核心原理:基于大肠杆菌生物素连接酶BirA的突变体BirA*(R11G突变)。该突变破坏了酶与生物素的稳定结合,使活化的生物素分子从酶上解离,随机标记邻近(约10nm范围内)蛋白质的暴露赖氨酸残基[1]。
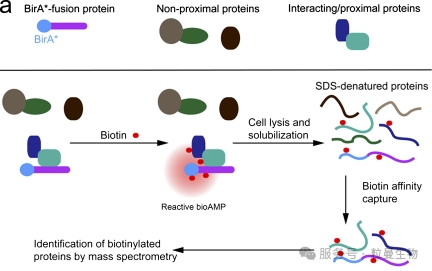
-
关键优势:
1)首次实现活细胞内非侵入性邻近标记,无需抗体或外源底物;
2)可靶向特定亚细胞结构(如核膜),揭示空间特异性相互作用。
-
局限性:
1)标记时间长(需孵育8-24小时),可能捕获非生理性相互作用;
2)酶分子量大(BirA*约35kDa),可能影响融合蛋白的定位或功能。
2. APEX(2013):毫秒级标记的 “闪电侠”
-
开发者:Alice Ting实验室
-
核心原理:工程化抗坏血酸过氧化物酶(APEX)在H₂O₂存在下,催化生物素-苯酚生成短寿命的生物素-苯氧自由基,快速标记邻近蛋白质的酪氨酸残基 [2]。

-
关键突破:
1)标记速度极快(仅需1-5分钟),可捕获瞬时相互作用;
2)成功绘制人类线粒体基质蛋白质组,解析线粒体钙单向转运蛋白的拓扑结构。
-
局限性:
1)依赖H₂O₂和生物素-苯酚,对细胞毒性较高,不适合类器官或体内研究[3]
2)灵敏度较低,需高表达量才能获得足够信号。
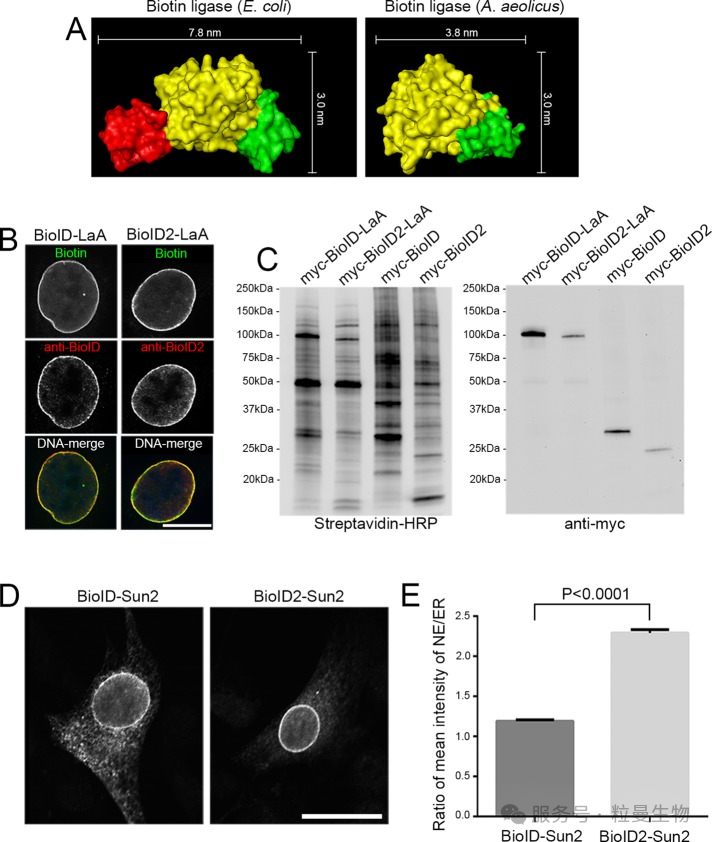
3. BioID2(2016):更小、更快的 “优化版”
-
开发者:Roux实验室
-
核心改进:将大肠杆菌BirA*替换为嗜热菌Aquifex aeolicus的生物素连接酶,删除DNA结合域,仅保留催化结构域(233个氨基酸),大小比BioID减少40%。[4]
-
性能提升:
1)标记效率提高,所需生物素浓度降低;
2)融合蛋白定位更精准,减少对靶蛋白功能的干扰。
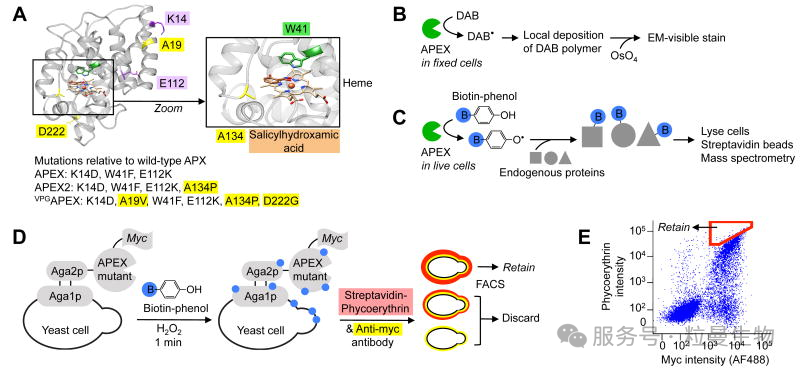
4. APEX2(2015):灵敏度革命
-
开发者:Alice Ting实验室
-
核心改进:通过定向进化获得突变体APEX2,解决野生型APEX折叠效率低、灵敏度不足的问题 [5]。
-
关键优势:
1)信号强度提升,低表达量下即可实现高效标记;
2)成功应用于突触间隙、内质网等复杂微环境的蛋白质组分析。
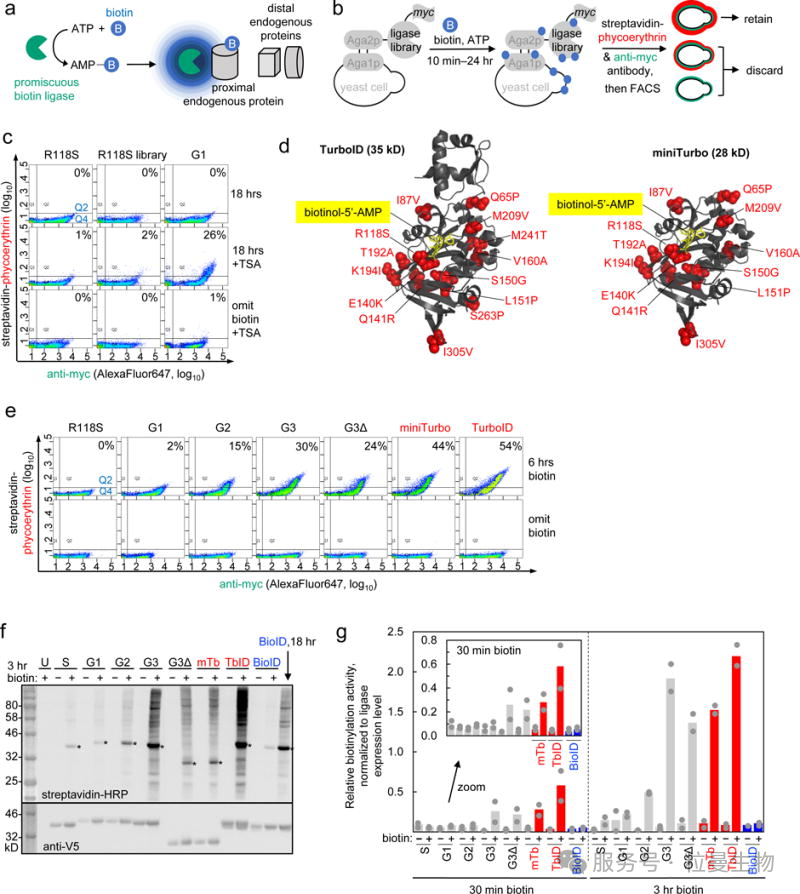
5. TurboID/miniTurbo(2018):兼顾速度与安全性的 “全能选手”
-
开发者:Alice Ting实验室
-
核心原理:通过酵母展示定向进化获得BirA突变体,TurboID(35kDa,15个突变)和miniTurbo(28kDa,13个突变,删除N端结构域)[6]。
-
技术飞跃:
1)标记时间缩短至10分钟,且无需H₂O₂,彻底解决毒性问题;
2)TurboID信号更强,miniTurbo背景标记更低,适合时空精准控制。
6. Split-BioID(2016):条件性相互作用研究工具
-
开发者:Schopp等
-
核心原理:将BirA*拆分为N端和C端片段,仅当融合的两个靶蛋白相互作用时,片段互补重构活性酶,实现“条件性标记”[7]。
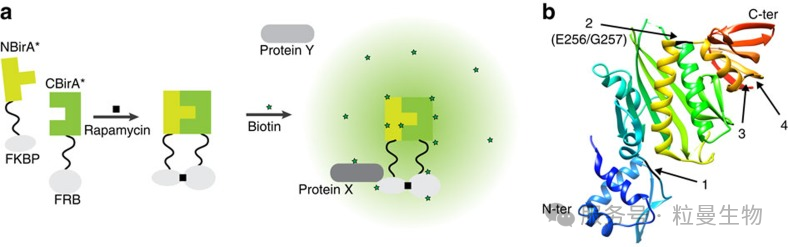
-
独特价值:
可同时验证蛋白质-蛋白质相互作用并捕获复合体中的其他成员,适用于动态相互作用研究。
7. RaPID(2018):RNA - 蛋白质相互作用的 “捕手”
-
开发者:Ramanathan 等
-
核心原理:基于枯草芽孢杆菌 BirA 突变体 BASU(动力学提升 1000 倍,信噪比提高 30 倍),融合 RNA 结合 motif,特异性标记与靶 RNA 结合的蛋白质[8]。
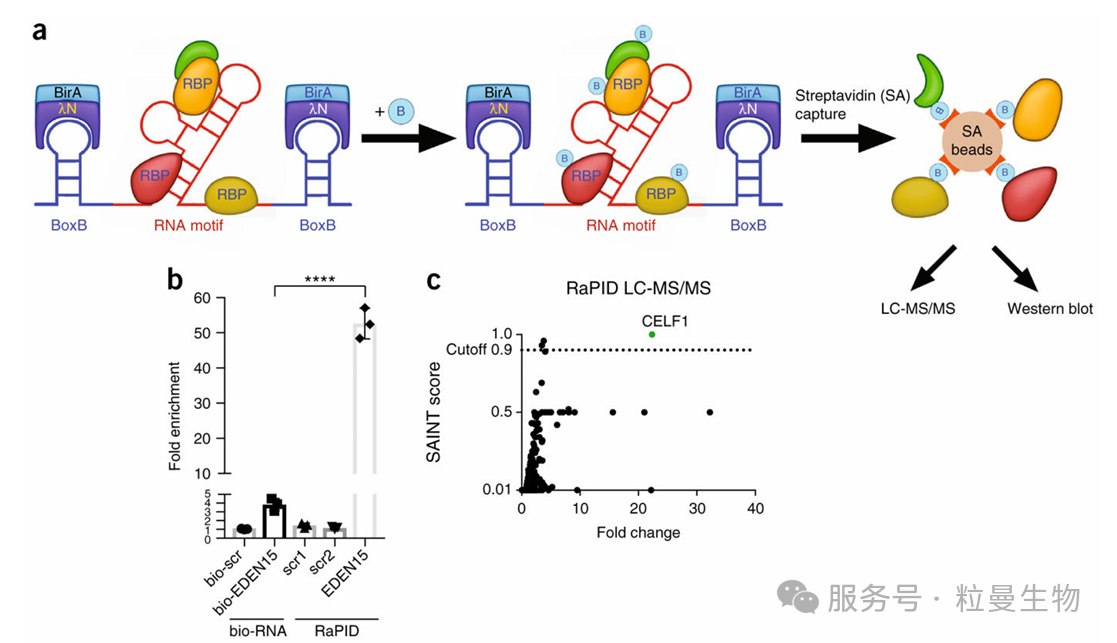
-
应用场景:解析 RNA 调控网络,如 lncRNA - 蛋白质复合体的鉴定。
8. Split-TurboID(2020):高特异性的“互作依赖型开关”
-
开发者:Alice Ting 实验室(2020年提出)
-
核心原理:将 TurboID 蛋白裂解为两个无活性的片段,分别融合到不同蛋白或定位元件上,只有在二者靠近时才重新组装,恢复生物素化活性,从而实现相互作用依赖性的邻近标记[9]。
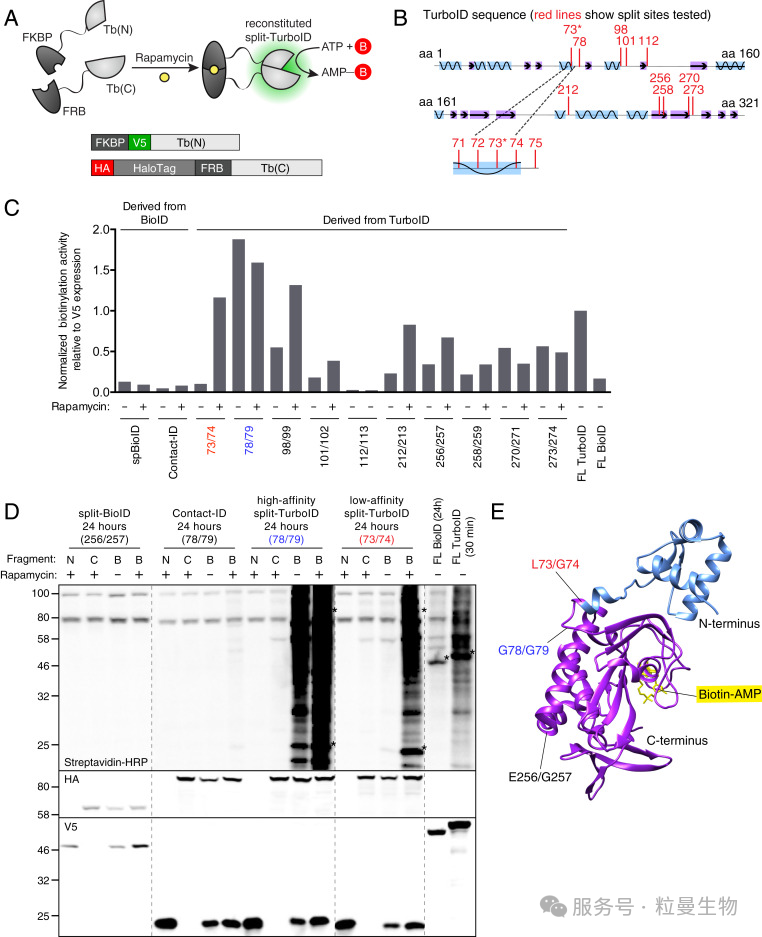
-
技术飞跃:
1)保持 TurboID 的快速标记能力(10分钟–1小时即可),同时显著降低背景信号;
2)通过互作依赖性恢复活性,实现更高的时空特异性;
3)适合研究蛋白–蛋白互作和条件依赖的动态接触。
二、技术对比:关键参数与适用场景
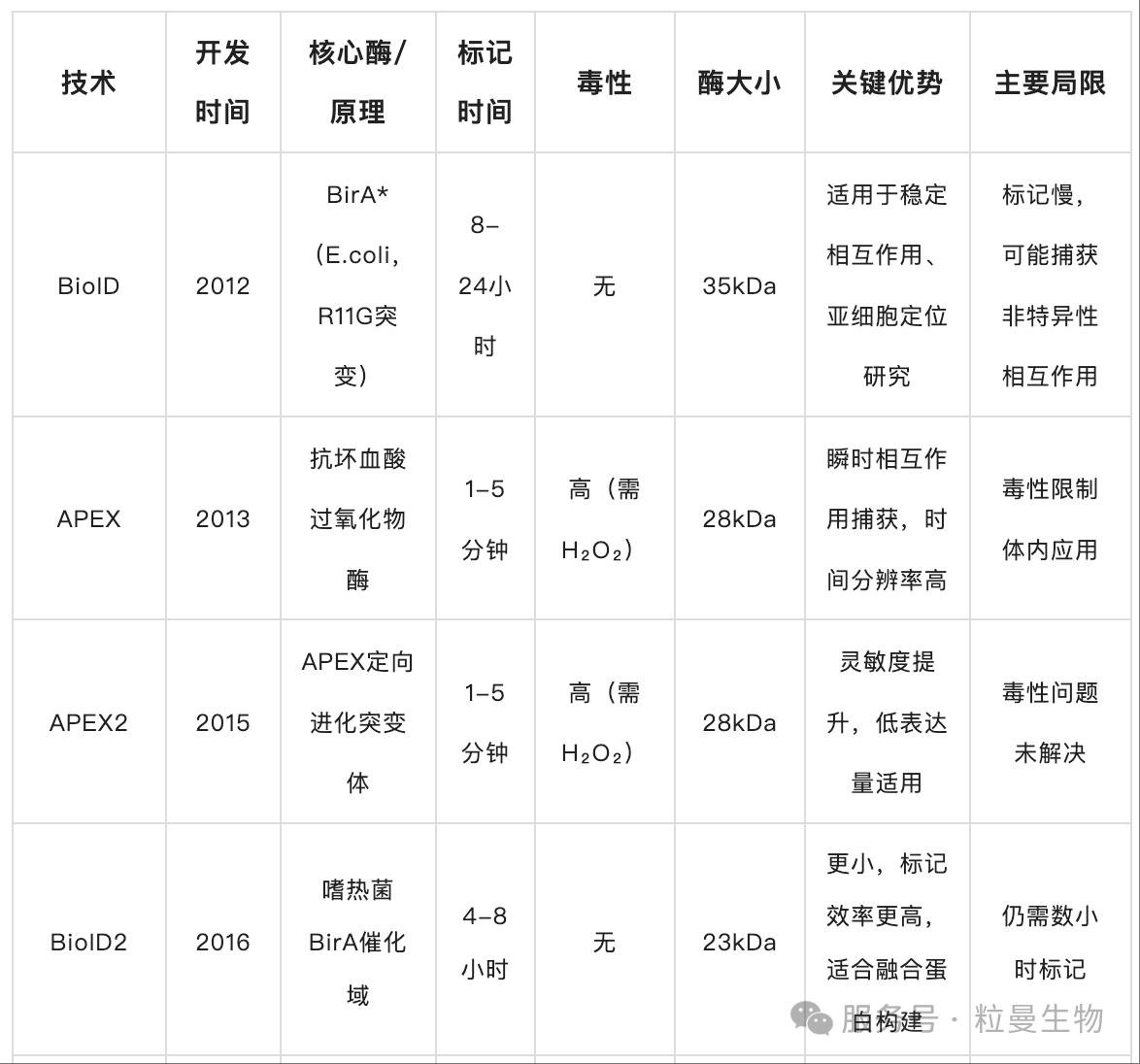
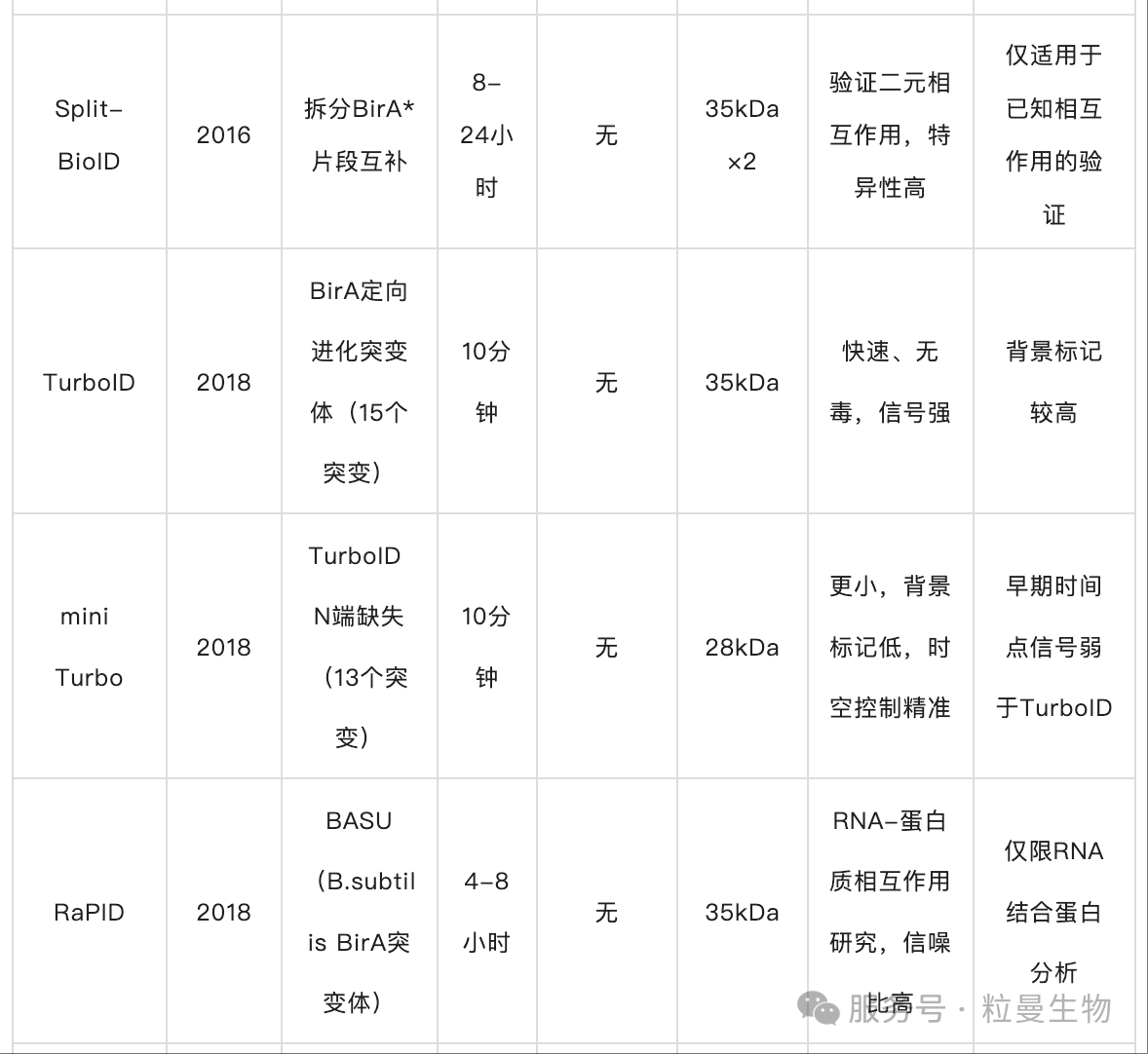

三、应用场景推荐
1. 亚细胞结构蛋白质组图谱
APEX技术凭借快速标记能力,成功绘制线粒体基质、突触后致密区等微环境的蛋白质组;TurboID则被用于神经元突起、初级纤毛等动态结构的实时相互作用捕获。
2. 疾病机制研究
BioID2助力发现核膜蛋白emerin的相互作用网络,揭示Emery-Dreifuss肌营养不良症的致病机制;RaPID鉴定lncRNA MALAT1的结合蛋白,为癌症转移研究提供新靶点。
3. 药物靶点发现
EXCELL在肿瘤微环境研究中,标记癌细胞与基质细胞的表面相互作用蛋白,为免疫检查点抑制剂开发提供候选分子。
四、未来展望:更高分辨率、更广适用性
邻近标记技术正朝着“时空精准化”、“多模态整合”方向发展:
-
更高分辨率:结合超分辨显微镜,实现纳米级空间定位的相互作用捕获;
-
更低毒性:开发无需H₂O₂的APEX变体,推动体内长期追踪研究;
-
多组学整合:与单细胞测序、空间转录组结合,解析细胞异质性中的蛋白质网络。
从BioID到TurboID,邻近标记技术的每一次突破都为生命科学研究打开新窗口。随着工具的不断优化,我们终将破解细胞内蛋白质相互作用的“暗物质”,为疾病诊断与治疗提供全新视角。
外展阅读:
文献解读|AirID:蛋白质相互作用研究的新篇章【收藏】(2020年)

参考文献
[1] Roux KJ, Kim DI, Raida M, Burke B. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. J Cell Biol. 2012 Mar 19;196(6):801-10.
[2] Rhee HW, Zou P, Udeshi ND, Martell JD, Mootha VK, Carr SA, Ting AY.Proteomic mapping of mitochondria in living cells via spatially restricted enzymatic tagging.Science.2013 Mar 15;339(6125):1328-1331.
[3] Che Y, Khavari PA. Research Techniques Made Simple: Emerging Methods to Elucidate Protein Interactions through Spatial Proximity. J Invest Dermatol. 2017 Dec;137(12):e197-e203.
[4] Kim DI, Jensen SC, Noble KA, Kc B, Roux KH, Motamedchaboki K, Roux KJ. An improved smaller biotin ligase for BioID proximity labeling. Mol Biol Cell. 2016 Apr 15;27(8):1188-96.
[5] Lam SS, Martell JD, Kamer KJ, Deerinck TJ, Ellisman MH, Mootha VK, Ting AY. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 2015 Jan;12(1):51-4.
[6] Branon TC, Bosch JA, Sanchez AD, Udeshi ND, Svinkina T, Carr SA, Feldman JL, Perrimon N, Ting AY. Efficient proximity labeling in living cells and organisms with TurboID. Nat Biotechnol. 2018 Oct;36(9):880-887.
[7] Schopp IM, Amaya Ramirez CC, Debeljak J, Kreibich E, Skribbe M, Wild K, Béthune J. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 2017 Jun 6;8:15690.
[8] Ramanathan M, Majzoub K, Rao DS, Neela PH, Zarnegar BJ, Mondal S, Roth JG, Gai H, Kovalski JR, Siprashvili Z, Palmer TD, Carette JE, Khavari PA. RNA-protein interaction detection in living cells. Nat Methods. 2018 Mar;15(3):207-212.
[9] Cho KF, Branon TC, Rajeev S, Svinkina T, Udeshi ND, Thoudam T, Kwak C, Rhee HW, Lee IK, Carr SA, Ting AY. Split-TurboID enables contact-dependent proximity labeling in cells. Proc Natl Acad Sci U S A. 2020 Jun 2;117(22):12143-12154.
2025 /
08-25
所属分类:
行业新闻
相关资讯—
2025-12-01
2025-12-01

